Fra Winston Ewerts nye fagfellevurderte artikkel om Avhengighetsmodellen
Brian Miller; 23. august 2023. Oversatt herfra
Winston Ewert har publisert en ny artikkel i tidsskriftet BIO-Complexity, og utvidet sin tidligere forskning relatert til mønsteret av likheter mellom arter. Han demonstrerte videre hvordan fordelingen av likheter passer hans avhengighetsgrafmodell bedre enn den vanlige felles-aner-modellen. Artikkelen kan lastes ned her. Jeg beskrev den overlegent forklarende kraften til Ewerts modell her. Hans originale bio-kompleksitetssrtikkel er her.
Bilde 1. Delfiner

Dawkins -testen
En av de sentrale spådommene for evolusjonsteori er at likheter mellom arter konsekvent bør peke på det samme evolusjonære treet basert på forutsetningen om felles aner. Forgreningspunktene i treet skal tilsvare den felles stamfaren til en gruppe organismer (f.eks. Pattedyr) som først skaffet seg alle egenskapene som er felles for den gruppen (f.eks. Kjertler for å produsere melk og tre bein i det indre øret).
Richard Dawkins uttalte at en pålitelig test av designhypotesen for livet, er om likheter er sterkt inkonsekvent med et enkelt tre. Data samlet inn i løpet av de siste tiårene har vist at fordelingen av likheter er langt mer inkonsekvent med felles aner enn noen evolusjonist forestilte seg, så det intelligente designrammen har bestått Dawkins -testen. Casey Luskin siterte en rekke studier som beskrev uoverensstemmelsene her.
Disse dataene undergraver kjernen av evolusjonsteorien. Nesten alle argumentene for de store evolusjonære fortellingene (f.eks. transformasjon av fisk til mennesker) hviler på antagelsen om at likheter pålitelig peker på felles aner. I mange grupper viser det høye nivået av inkonsekvens av likheter med ethvert evolusjonært tre, at denne grunnleggende antagelsen er feilaktig.
Spesielt problematisk, er stedene der evolusjonister mest krever pålitelige data, er der dataene er mest inkonsekvente. For eksempel er et sentralt ikon for evolusjon av "hvalserien" som består av fossiler som angivelig illustrerer et landpattedyr som trinnvis transformerer seg til et fullt vannlevende pattedyr. Dyrene i serien antas ikke direkte å ha avstammet fra, eller utviklet seg til hverandre, men likhetene deres blir tolket som at de har stammet fra dyr som er en del av en forfedre-nedsatt serie som omfatter land-sjø-overgangen.
Avhengighetsgrafmodell
Derimot passer mønsteret av likheter designbaserte modeller, som jeg tidligere beskrev her. For å oppsummere bruker menneskelige ingeniører designmoduler, for eksempel motorer, i forskjellige kreasjoner for å oppfylle felles mål. På samme måte er likheter mellom arter ofte ikke i samsvar med et evolusjonært tre, men de ser ut til å være implementert i forskjellige skapninger for et felles formål. Ewerts rammeverk er den første som identifiserer moduler basert på proteiner.
I Ewerts første artikkel identifiserte han moduler som sett med proteiner fra forskjellige familier. Noen moduler er avhengige av mindre moduler, og de mindre modulene brukes til tider av flere større moduler (figur 1). Ewert demonstrerte kvantitativt at proteindataene passer langt bedre til avhengighetsgrafmodellen enn felles avstamnings-modell.
Fig 1. Del av avhengighetsgraf for proteiner
Anve ndelse på proteinsekvenser
ndelse på proteinsekvenser
I Ewerts nylig publiserte artikkel identifiserte han ikke moduler som sett med distinkte proteiner. I stedet fokuserte han på variasjoner i proteinet prestin mellom en rekke pattedyrarter, inkludert de som er i stand til ekkolokalisering. Proteinet spiller en kritisk rolle i pattedyrhørselen ved å tjene som et motor protein i de ytre hårcellene. Versjonene av proteinet som støtter ekkolokalisering flaggermus, hvaler og delfiner viser de samme aminosyreendringene sammenlignet med versjonene i pattedyr som mangler ekkolokalisering. Endringsmønsteret er ikke enkelt. Ulike par pattedyr deler lignende aminosyreforandringer. Konstruerte evolusjonstrær for pattedyr basert på felles aner mister denne informasjonen, mens Ewerts modell fremhever den.
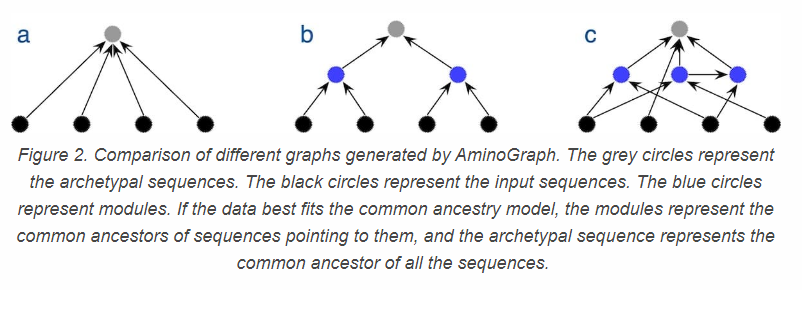
For å visualisere sammenhengen, opprettet Ewert et program kalt Aminograph som identifiserer moduler som spesifikke aminosyreendringer fra en arketypisk sekvens som er en representativ versjon av proteinet. En modul avhenger av en annen hvis den inkluderer sistnevnte endringer, pluss ytterligere endringer. Programmet tar som inngang aminosyresekvenser for det samme proteinet i forskjellige arter, og det skaper fra disse dataene, den mest konsistente grafen for modulavhengigheter.
Programmet viser sett med sekvenser som ikke er relatert, da hver inngangssekvens direkte kobler til den arketypiske sekvensen (figur 2A). Den viser data som best passer til en felles avstamnings-modell som et standard evolusjons tre (figur 2B), og den viser data som tilsvarer moduler med komplekse sammenhenger som en avhengighetsgraf (figur 2C). Ewert testet programmet på simulerte data som tilsvarer ikke -relaterte sekvenser, sekvenser koblet med vanlige aner og sekvenser koblet gjennom en spesifikk avhengighetsgraf. I begge tilfeller identifiserte programmet riktig struktur og identifiserte de fleste av modulene og deres sammenheng og bekreftet slik påliteligheten til programmet.
Fig 2. Sml. av grafer v. Aminograph
Resultater for prestin-proteiner
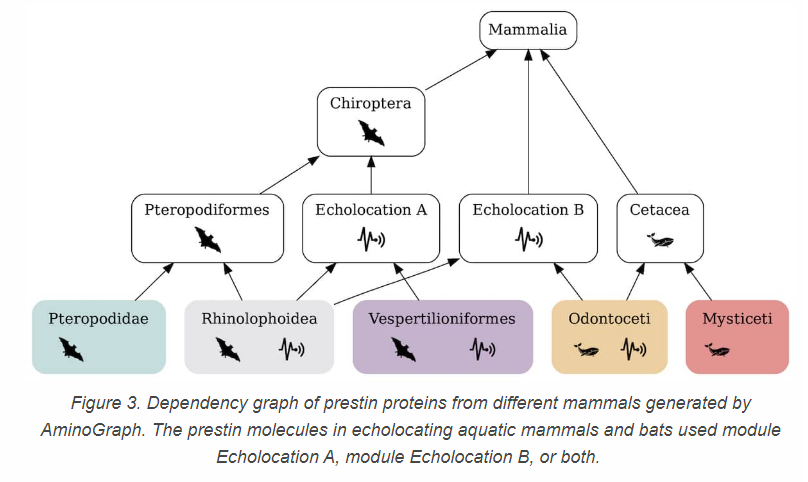
Ewert kjørte Aminograph på sekvensene av prestinproteiner fra arter av ekkolosering av flaggermus, flaggermus uten ekkolokalisering, ekkolokaliserende hvaler, hvaler uten ekkolokasjon og andre pattedyr uten ekkolokasjon. Den genererte avhengighetsgrafen avslørte en nøkkelinnsikt i hvordan prestin-proteinene ble modifisert, for å svare på de høye lydfrekvensene generert i ekkolokalisering. Reengineering i hvert ekkolokaliserende pattedyr benyttet en kombinasjon av to moduler merket ekkolokalisering A og ekkolokalisering B (figur 3).
Fig 3. Avhengighetsgraf for Prestin-protein
Fremtidig forskning
Dette resultatet fører naturlig til forskning på effekten av aminosyre-modifikasjoner. Designrammen spår at endringer koordinerer i hver modul for å støtte ekkolokasjon på en måte som best tjener behovene til arten som bruker den. En annen forventning er at prestinmodulene må koordineres med andre moduler for ekkolokalisering, for å fungere på et nivå som vil være til fordel for besitteren. Denne prediksjonen virker i samsvar med de mange likhetene mellom å ekkolokaliserende pattedyr, både i deres genetikk (her, her) og deres overordnede designlogikk (her, her).
Derimot gir standard evolusjonsramme ingen innsikt i hvordan ekkolokalisering implementeres hos forskjellige pattedyr. Det ofte aksepterte evolusjonære treet krever at ekkolokalisering har utviklet seg uavhengig tre ganger som illustrert i Ewert (2023), figur 1 . Likevel er maksimal mulig tid for utviklingen av et fullt vannlevende pattedyr eller en flaggermus utilstrekkelig for mer enn to koordinerte mutasjoner skal kunne vises . Ethvert evolusjonsscenario for ekkolokalisering vil kreve langt mer enn to koordinerte mutasjoner (her, her), så evolusjonen klarer ikke å forklare denne egenskapens opprinnelse endog én gang, enn si flere ganger.
Ewerts modell kan brukes på flere andre proteiner i ekkolokalisering og i andre biologiske systemer for å få lignende innsikt. Den representerer et verdifullt verktøy i den utviklende teorien om biologisk design, som til slutt skal erstatte fylogenetiske analyser.

Brian Miller
Forskningskoordinator, Center for Science and Culture
Dr. Brian Miller er forskningskoordinator for Center for Science and Culture ved Discovery Institute. Han har en B.S. i fysikk med en lavere grad i prosjektering fra MIT og en doktorgrad. i fysikk fra Duke University. Han taler internasjonalt om temaene Intelligent Design og effekten av livssyn på samfunnet. Han har også konsultert om organisasjonsutvikling og strategisk planlegging, og han er en teknisk konsulent for Ideashares, en virtuell inkubator dedikert til å bringe innovasjon til markedet.
Oversettelse og bilder ved Asbjørn E. Lund


 ndelse på proteinsekvenser
ndelse på proteinsekvenser

